水に含まれる環境DNAから「どんな魚」が「どれだけいるか」を同時に推定
-定量的な魚類群集モニタリングを容易に実現-
山口大学環境DNA研究センターの辻冴月(つじさつき)学術研究員(現・京都大学)と赤松良久(あかまつよしひさ)教授、福岡工業大学の乾隆帝(いぬいりゅうてい)教授らの研究グループは、九州・中国地方の複数河川における大規模な調査により、水に含まれる魚類の環境DNAを定量的環境DNAメタバーコーディングにより定量的・網羅的に分析することで、「どんな魚類」が「どれだけ生息しているか」を同時に推定できることを明らかにしました。本成果は、2022年12月13日に国際学術誌「Scientific Reports」に掲載されました。
研究成果のポイント
- 一般的に用いられる環境DNAメタバーコーディングでは、分析上の制限により、種の存在を検出できるものの、それらの量を定量的に評価することは困難でした。
- 既知濃度の内部標準DNAを試料に添加することで、メタバーコーディングにおける(半)定量評価を可能にする定量メタバーコーディング法(以下、qMiSeq法)がUshio et al. (2018)により開発されました。
- 魚類の環境DNAメタバーコーディングにqMiSeq法を適用し、推定された環境DNA濃度が、調査地に生息する各種の個体数や生物量を反映するか検証しました。
- 推定された各種の環境DNA濃度は、採水調査時に電気ショッカーを用いて捕獲された各魚種の個体数および生物量を反映していることが示されました。
- 環境DNA定量メタバーコーディングは生物多様性の定量的なモニタリングを容易に実現する有用性の高い手法と言えます。
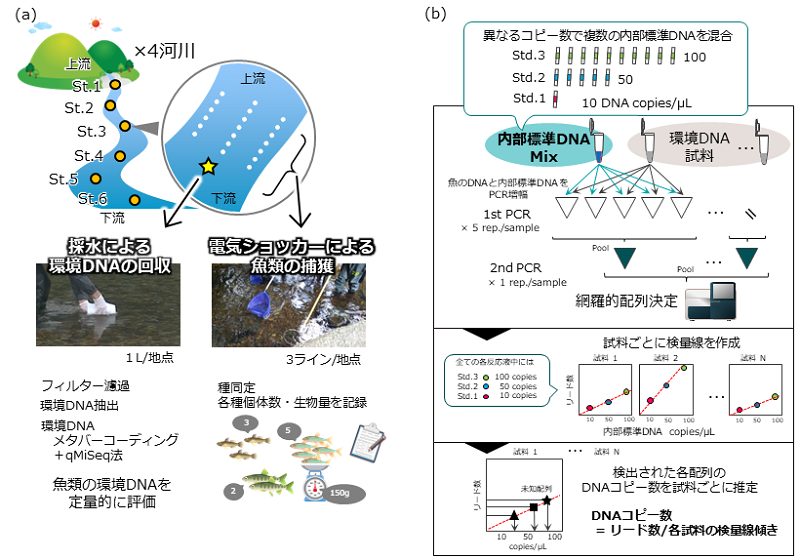 図1. (a) 野外調査の概略 (b) qMiSeq法の分析手順
図1. (a) 野外調査の概略 (b) qMiSeq法の分析手順
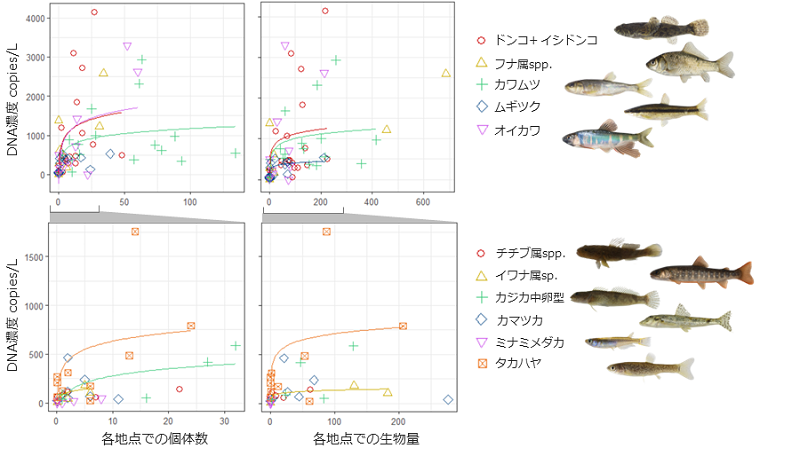 図2. 地点間で検出頻度の高かった11種について、定量化された各種DNA濃度と捕獲データ(個体数および生物量)との関係。
図2. 地点間で検出頻度の高かった11種について、定量化された各種DNA濃度と捕獲データ(個体数および生物量)との関係。
負の二項分布に基づく一般化線形モデル(GLM)の結果が有意(p<0.05)な種のみ、関係を実線で示した。
発表論文の概要
- 研究論文名:Quantitative environmental DNA metabarcoding shows high potential as a novel approach to quantitatively assess fish community
- 著者:辻冴月(京都大学大学院理学研究科、山口大学大学院創成科学研究科)*、乾隆帝(福岡工業大学社会環境学部)、中尾遼平(山口大学大学院創成科学研究科)、宮園誠二(山口大学大学院創成科学研究科)、齋藤稔(山口大学大学院創成科学研究科、国立研究開発法人 国際農林水産業研究センター)、河野誉仁(国立研究開発法人 土木研究所)、赤松良久(山口大学大学院創成科学研究科)
*責任著者 - 公表雑誌:Scientific Reports
- 公表日:2022年12月13日
- DOI:10.1038/s41598-022-25274-3
謝辞
本研究は、下記の研究助成を受けて行いました。
・公益財団法人河川財団 河川基金(2019-5211-030)
・山口大学YUプロジェクト
・日本工営共同研究講座

